Dataset utilisé
Special thanks to Samir Bhattarai for this amazing dataset !
Pour cette activité de détection des maladies des bois de la vigne, nous utiliserons une partie du dataset : les répertoires concernant les feuilles de vigne.
Un répertoire contient des photos de feuilles de vigne saines, trois autres répertoires contiennent des photos de feuilles de vigne atteintes de maladies.
Cette activité est réalisée avec deux répertoires : « healthy » et « ESCA ».
Découvrir le Transfer Learning⚓
Principe du transfer learning
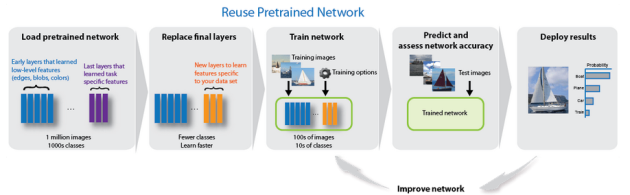
Le transfer learning consiste à partir d'un réseau de neurones déjà entraîné à reconnaître des images, et à le réentraîner sur des images. L'avantage est que le réseau est déjà existant (couches de neurones de différents types reliées entre elles), avec des paramètres fonctionnels pour certaines images.
Il sera beaucoup plus facile d'entraîner ce réseau qui devra recalculer et modifier ses paramètres que de créer complètement un nouveau réseau de neurones.
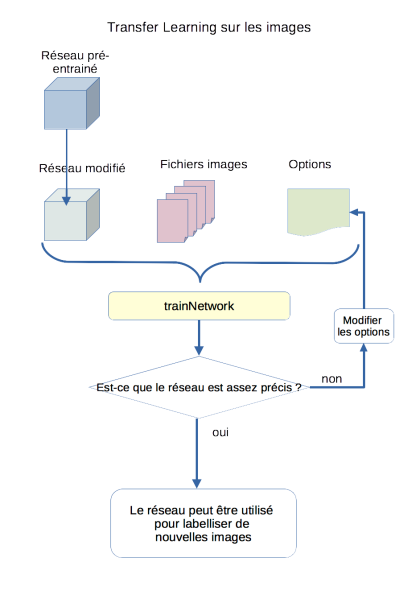
Méthode : Étape 1 : Modifier un réseau existant
Matlab permet de charger un réseau pré-entraîné existant, et de le modifier pour l'adapter à nos besoins. Pour cette activité nous utiliserons le cnn GoogleNet.
La modification se limite à modifier deux couches
classification layer : remplacer la couche existante par une nouvelle couche de classification vierge avec en 'output size' le nombre de classes à identifier
output layer : remplacer la couche existante par une nouvelle couche output vierge
Couche « fully connected » : chaque neurone est connecté à tous les neurones de la couche suivante
Méthode : Étape 2 : Préparer les fichiers images
Les images sont le plus souvent organisées dans une arborescence. Sous Matlab nous pourrons utiliser les "imageDatastore", une variable qui regroupe les images situées dans un répertoire (avec ou non des sous-répertoires).
L'utilisation des imageDatastore présente plusieurs avantages intéressants
si il faut retailler les images pour les adapter au cnn, le datastore permet de retailler automatiquement chaque image à la volée.
les images sont tirées une par une du datastore de façon transparente au moment du traitement, ce qui évite de charger toutes les images en mémoire
Matlab sait répartir les images pour avoir un ensemble pour le training et un ensemble pour le test
Méthode : Étape 3 : Définir les options d'entraînement
On peut très bien lancer l'entraînement avec les options par défaut au début.
Parmi les options :
l'algorithme de descente de gradient (sgdm, adam, rmsprop)
le nombre maximum d'époques
la taille des min-batches
...
Personnellement j'ai eu de bien meilleurs résultats avec l'algorithme « sgdm » qu'avec « adam » qui est mis par défaut.
Référence : Documentation sur les options d'entraînement des cnn
Méthode : Étape 4 : Entraînement et évaluation du réseau de neurones
Le temps de calcul d'un entraînement peut être très long (plusieurs heures). Il faut donc penser à enregistrer le nouveau réseau en fin de calcul !
Il est possible d'entraîner le réseau sur une sous-partie des images et/ou en limitant le nombre d'époques, pour valider les options, avant de lancer l'entraînement global.
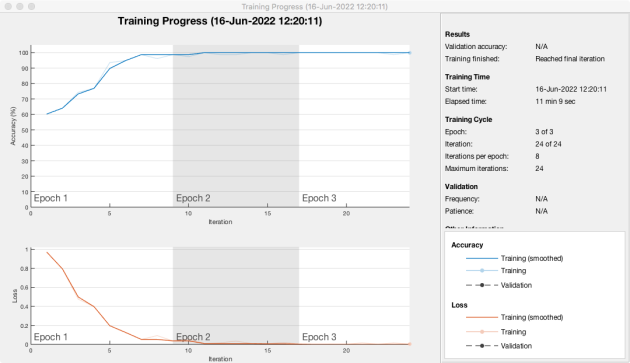
Ici, le dataset est modeste et l'entraînement prend entre 6 et 12 minutes.
Après l'entraînement, le réseau peut être utilisé, on cherchera à contrôler la matrice de confusion pour valider la précision du réseau.
Entraînement d'un réseau neuronal en ligne de commande⚓
Conseil : Matlab Live Editor
Le live editor de Matlab permet d'enregistrer le code ainsi que le résultat de l'exécution, et permet également de créer des sections qui peuvent être exécuter de façon indépendante.
Le live editor permet également d'améliorer la lisibilité avec des blocs de texte insérés dans les sections.
Le live editor permet de créer des sections de code, et d'enregistrer les résultats de l'exécution des sections.
Étape 1 : Modifier un réseau existant
% on réutilise GoogleNetnet = googlenet
lay = net.Layers
% affichage de la taille d'entrée dans la command window (facultatif)inLayer = lay(1)
inSize = inLayer.InputSize
% création du nouveau réseau en modifiant deux coucheslgraph = layerGraph(net);
% taille 2 car le cnn doit reconnaître 2 classes d'imagesnewFc = fullyConnectedLayer(2,"Name","new_fc");
lgraph = replaceLayer(lgraph,"loss3-classifier",newFc)
newOut = classificationLayer("Name","new_out");
lgraph = replaceLayer(lgraph,"output",newOut)
Attention : Classification layer / Output layer
La couche de classification est une des dernières couches : elle attribue un score à chaque classe pour l'image traitée. Son type est "fully connected layer" car ce sont des neurones connectés.
La classe de sortie (output layer) traite les scores, les traduit en probabilités pour chaque classe, et donne en sortie la classe la plus probable (le label). Son type est sous Matlab est "Classification Output"
Il y a parfois ambiguïté entre la "classification layer" et la "output layer" car Matlab a attribué le type 'Classification Output' à la couche de sortie.
Étape 2 : Préparer les fichiers images
% création du datastore : les labels sont les noms des répertoiresimds = imageDatastore("grape2classes/", "IncludeSubfolders",true, "LabelSource","foldernames")
imds.Labels
% répartition lot d'entraînement / lot de test[trainAllImgs, testAllImgs] = splitEachLabel(imds, 0.7, "randomized")
% les images seront retaillées à la volée par le cnn, sans modification de l'image originale% mise à la taille de la taille d'entrée du cnntrainds = augmentedImageDatastore([224 224],trainAllImgs);
testds = augmentedImageDatastore([224 224],testAllImgs);
Etape 3 : Définir les options d'entraînement
Algorithme = sgdm
MaxEpochs = 3 pour limiter le temps de calcul (C'est suffisant ici, on pourrait mettre aussi 5 si on a plus de temps)
MiniBatchSize = 78
J'ai pris des mini-batches de 78 images car j'ai 624 images dans le lot d'entraînement (624 = 8x78)
Shuffle = every epoch : le réseau mélange les images au début de chaque époque pour que les mini-batches ne soient pas constitués de façon identique
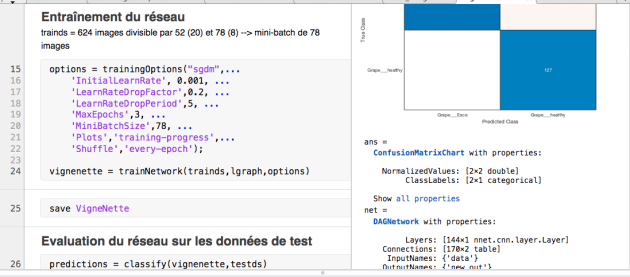
options = trainingOptions("sgdm",...
'InitialLearnRate', 0.001, ...
'LearnRateDropFactor',0.2, ...
'LearnRateDropPeriod',5, ...
'MaxEpochs',3, ...
'MiniBatchSize',78, ...
'Plots','training-progress',...
'Shuffle','every-epoch');
Pour plus de détails : Doc Matlab : trainingOptions
Etape 4 : Training et évaluation
% entraînement du cnn, qui sera chargé dans la variable grapenetvignenette = trainNetwork(trainds,lgraph,options)
% enregistrement dans un fichier .mat pour pouvoir réutiliser le cnnsave VigneNette
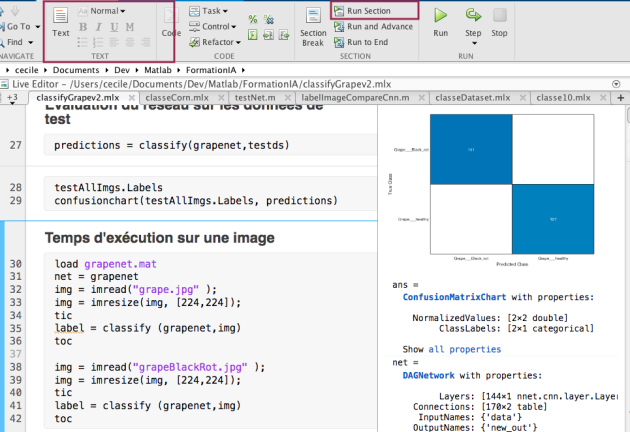
predictions = classify(vignenette,testds)
testAllImgs.Labels
% trace la matrice de confusionconfusionchart(testAllImgs.Labels, predictions)
% test sur des images et calcule le temps d'exécution (affiché dans la command window)img = imread("grape.jpg" );
img = imresize(img, [224,224]);
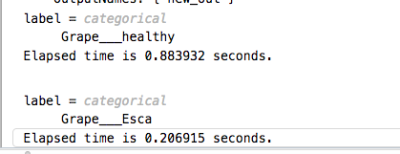
ticlabel = classify (vignenette,img)
tocimg = imread("grapeBlackRot.jpg" );
img = imresize(img, [224,224]);
ticlabel = classify (vignenette,img)
tocEntraîner googleNet à reconnaître les feuilles de vigne porteuses de maladie (ESCA)
Question⚓
Ecrire un live script Matlab pour réentraîner le réseau googleNet à reconnaître les feuilles de vigne porteuses de maladies (ESCA)
Solution⚓
Voir sur Github SI-IA-VigneNette le fichier vigneNette.mlx
Ici on voit que 3 époques peuvent suffire pour un bon résultat (pour limiter le temps de calcul).
On pourrait aussi augmenter le nombre d'époques
Le problème du temps de calcul⚓
Entraînement sur tout le dataset
La fonction "train" est très gourmande en ressources et en particulier en temps de calcul. L'utilisation de GPU peut permettre de gagner du temps. Par rapport aux contraintes horaires, on peut lancer un entraînement sur une partie du dataset pour faire des tests (algorithmes, options, nb d'epochs, mini-batch etc.) avant de lancer le transfer learning sur toute la base des images.
Méthode : Limiter le volume des images d'entraînement
Ici le dataset est réduit, ce qui permet d'avoir des temps de calcul minime. Si on travaille sur un dataset plus important, on peut faire des tests sur un volume plus réduit pour valider les paramètres avant de lancer les calculs sur tout le dataset.
Méthode :
Limiter les images d'entraînement (ex : 500 pour le training, 150 pour le test)
Étudier les résultats (matrice de confusion, précision, temps d'exécution d'une classification)
Modifier les options d'entraînement (algorithme, nombre d'époques...) et relancer l'entraînement
Quand les paramètres permettent d'approcher une bonne précision, on peut conserver ces options et lancer le training sur tout le dataset
Complément : utiliser un sous-ensemble d'images et algorithmes⚓
Se limiter à un sous-ensemble des dossiers (exemple avec la vigne : 2 répertoires)
Cela permet de travailler sur un sous-ensemble d'images pour valider l'algorithme et certains paramètres. Quand cela donne des bons résultats, le calcul peut ensuite être lancé sur tout le dataset.
Utilisation de la fonction "subset"
Proposition de solution pour créer les deux sous-ensembles d'images : les images doivent être choisies de façon aléatoire et de façon équitable depuis tous les dossiers.
% Creation des datastores pour training et test du cnnimds = imageDatastore("img", "IncludeSubfolders",true, "LabelSource","foldernames")
imds.Labels
[trainAllImgs, testAllImgs] = splitEachLabel(imds, 0.7, "randomized")
%pour travailler sur une partie des images et pas sur la totaliténFiles = length(trainAllImgs.Files);
RandIndices = randperm(nFiles); %mélange aléatoire des indices des fichiers images
indices = RandIndices(1:500) % on ne prend que les 500 premières images (après mélange)
trainPartImg = subset(trainAllImgs,indices) %création d'un datastore extrait
%idem pour le datastore dédié au test du cnnnFiles = length(testAllImgs.Files);
RandIndices = randperm(nFiles);
indices = RandIndices(1:200)
testPartImg = subset (testAllImgs, indices)
% si besoin de modifier la taille de l'image pour l'adaptertrainds = augmentedImageDatastore([224 224],trainPartImg);
testds = augmentedImageDatastore([224 224],testPartImg);
Les options de la fonction "train"
Référence : Doc Matlab : trainingOptions
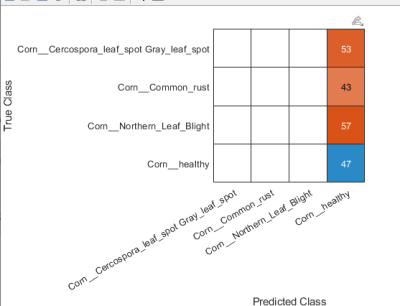
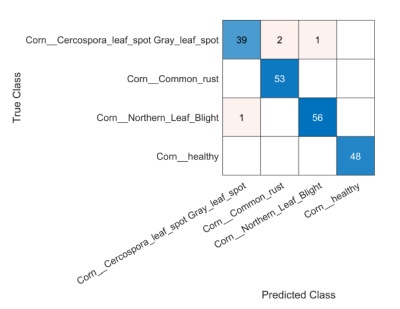
Choix de l'algorithme "solver" : la matrice de confusion
Pour mesurer l'importance du choix de l'algorithme, voici pour info deux tests effectués sur un dataset plus complet : l'un avec l'algorithme "adam" et l'autre avec « sgdm »
L'algorithme « sgdm » a donné de biens meilleurs résultats c'est pourquoi je l'ai utilisé pour VigneNette.